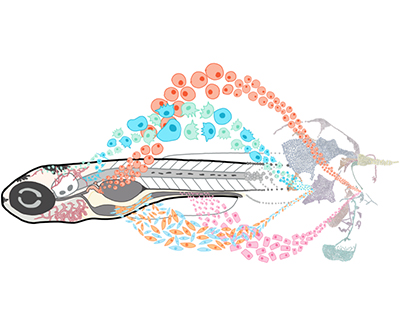
Ilustración del concepto de análisis unicelular durante el desarrollo temprano del pez cebra.
Crédito: J. Farrell, NICHD/NIH
Los investigadores científicos se basan en organismos modelo como el pez cebra para determinar cómo maduran los embriones a partir de una sola célula hasta desarrollar distintos tipos de células, tejidos y órganos. Muchos de los programas de expresión génica que dirigen el crecimiento embrionario son similares en los peces y las personas, y los hallazgos en el pez cebra pueden servir de base para estudios sobre el desarrollo y las enfermedades humanas.
En un estudio reciente del Laboratorio Farrell ![]() , los investigadores científicos utilizaron un método denominado secuenciación unicelular del ARN para identificar los programas de expresión génica que están activos en los primeros cinco días de desarrollo del pez cebra. En conjunto, este recurso sirve de atlas genético del desarrollo temprano
, los investigadores científicos utilizaron un método denominado secuenciación unicelular del ARN para identificar los programas de expresión génica que están activos en los primeros cinco días de desarrollo del pez cebra. En conjunto, este recurso sirve de atlas genético del desarrollo temprano ![]() .
.
- El atlas describe casi 490,000 células de 62 puntos temporales durante las primeras 120 horas después de la fecundación, con un promedio de 8,621 transcripciones y 1,745 genes detectados por célula.
- A continuación, el equipo del estudio clasificó estos datos en distintos tipos y estados celulares e identificó más de 200 tipos celulares presentes durante este período del desarrollo.
- Para resaltar la utilidad del atlas, el equipo se centró en el desarrollo de células poco estudiadas, incluidas las células intestinales llamadas células BEST4+, que podrían estar relacionadas con enfermedades gastrointestinales y cáncer en las personas. Se sabe poco sobre cómo se desarrollan estas células porque están ausentes en otros organismos modelo comunes, como los ratones.
- Utilizando el atlas, el equipo predijo computacionalmente el programa de desarrollo completo de las células BEST4+, incluidas las señales que inician el desarrollo de las células y los factores de transcripción que llevan a cabo el proceso. Estos hallazgos pueden evaluarse en organismos modelo o muestras clínicas para comprender mejor el papel de las células BEST4+ en las enfermedades humanas.
- El atlas, denominado Daniocell, es de acceso público para la comunidad de investigación en general en https://daniocell.nichd.nih.gov
 . Además, también están disponibles imágenes microscópicas relacionadas con el estudio
. Además, también están disponibles imágenes microscópicas relacionadas con el estudio 
.
Entre los coautores del documento del NICHD se encuentran Abhinav Sur, Paulina Capar, Morgan Kathleen Prochaska, Gennady Margolin y Jeffrey A. Farrell.
Obtenga más información sobre el Grupo de Afinidad de Regulación y Desarrollo Celular: https://www.nichd.nih.gov/about/org/dir/affinity-groups/CRD ![]()
 ARRIBA
ARRIBA